Reprogramación de secuencias de comandos de monocitos en COVID-19
En un estudio reciente publicado en bioRxiv* Servidor, los investigadores examinaron la función de diferenciación del grupo 14 positivo (CD14).+) monocitos en pacientes con enfermedad por coronavirus leve o moderada 2019 (COVID-19).
La infección respiratoria por COVID-19 causa síntomas leves o ningún síntoma en la mayoría de las personas. Sin embargo, la enfermedad de moderada a grave se observa en aproximadamente el 15 % de los casos, mientras que la enfermedad grave se observa en el 5 % del total de casos. Durante la fase de infección aguda, las células mieloides, como los monocitos/macrófagos, se enriquecen ampliamente en el pulmón de los pacientes con COVID-19.
Los monocitos son células inmunitarias innatas, difusivas y fagocíticas involucradas en la detección de patógenos y la activación de la inmunidad innata y adaptativa en respuesta a la infección viral. Los monocitos se diferencian en macrófagos y células dendríticas (DC) en los tejidos afectados y contribuyen a la eliminación de patógenos y la regeneración de tejidos. Varios estudios informaron respuestas innatas desreguladas contra el SARS-CoV-2, con células mieloides que muestran una expresión alterada del homotipo del antígeno leucocitario humano (HLA)-DR.
Estudio: reprogramación transcripcional de funciones inmunitarias innatas a una firma protrombótica al detectar el SARS-CoV-2 por monocitos en COVID-19. Haber de imagen: Mohamed Oweidat/Shutterstock
Estudio y resultados
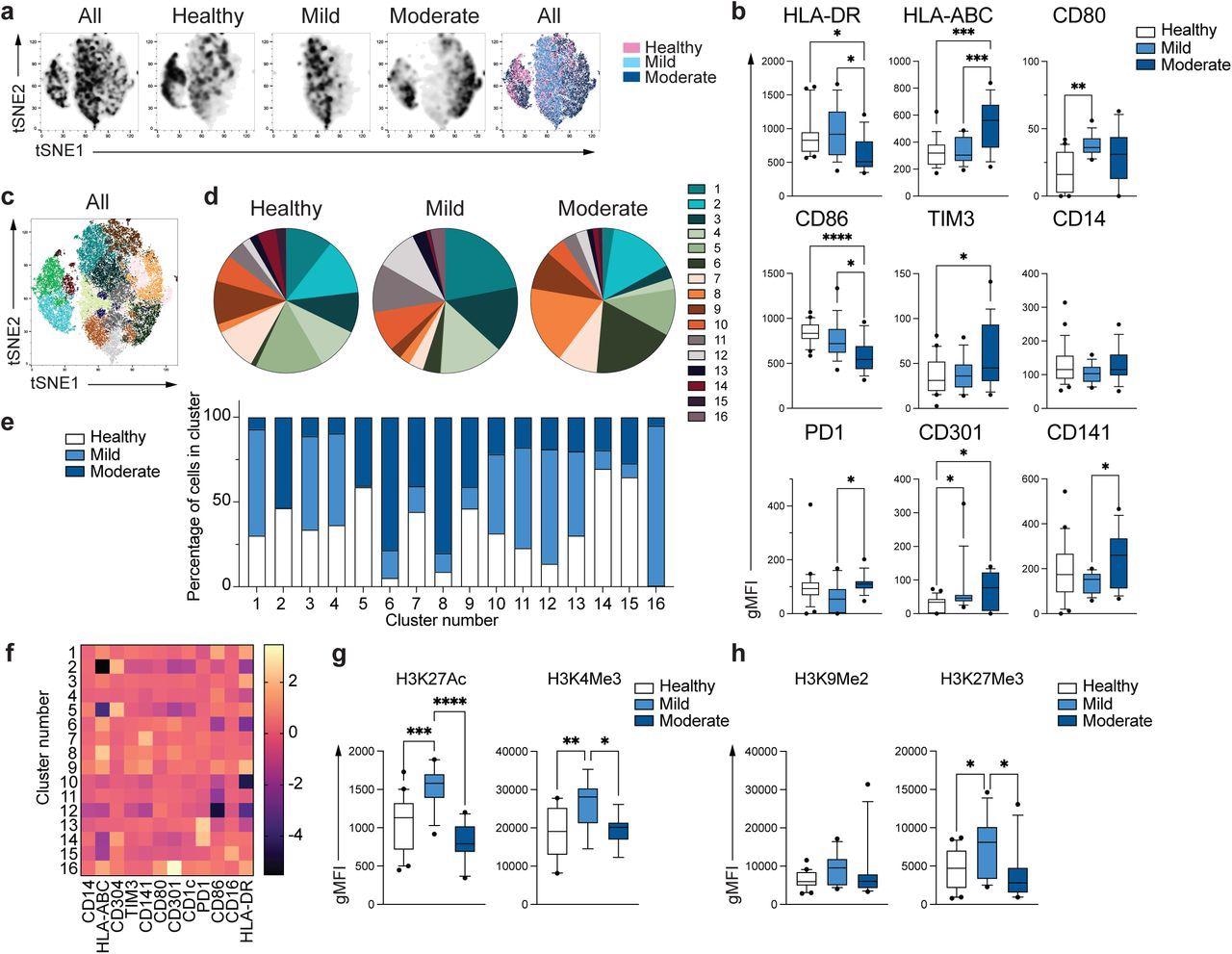
En este estudio, los investigadores evaluaron las características fenotípicas y funcionales del clásico (CD14.4)+) monocitos en pacientes con COVID-19 en comparación con controles sanos en este estudio. La citometría de flujo de alta dimensión evaluó las moléculas del complejo principal de histocompatibilidad (MHC), los receptores de costo y los inhibidores. Aunque algunos fenotipos globales se superpusieron en los tres grupos, solo las células de los controles sanos se diferenciaron de los pacientes con COVID-19 leve y moderado.
Los monocitos de las muestras de COVID-19 leve mostraron una expresión baja de HLA-DR, pero la expresión de HLA-ABC fue alta en relación con las muestras de control y de COVID-19 leve. Los monocitos de muestras leves de COVID-19 mostraron una expresión reducida de CD86, un receptor de costo, con aumentos en los receptores inhibidores: inmunoglobulina de células T, receptores de dominio de miosina que contiene 3 (TIM-3) y muerte programada 1 (PD-1) .
Los investigadores observaron 16 subpoblaciones unicelulares (grupos). La distribución de células en los tres grupos difirió en cada grupo, revelando grupos y monocitos distintos en pacientes con COVID-19 leve y moderado. Los patrones de activación o represión de la expresión génica se exploraron mediante el estudio de marcas epigenéticas asociadas con la transcripción activa como H3K27Ac (acetilación de histonas) y H3K4Me3 (metilación de histonas) y la represión de genes como H3K9Me2 y H3K27Me3.
El fenotipo único de los monocitos COVID-19.a. Gráficos de tSNE obtenidos de una muestra en serie que consiste en PBMC de n = 15 individuos sanos, n = 15 moderados y n = 15 pacientes moderados con COVID-19. B. Gráficos de caja y bigotes que resumen la mediana de gMFI para los receptores analizados. El fondo se extiende desde 25y a 75y el percentil y los bigotes disminuyen a 10y Porcentaje y hasta 90y porcentaje. Los puntos por debajo y por encima de los bigotes se trazan como puntos individuales (n = 25 sanos, n = 15 leves, n = 17 individuos con COVID-19 moderado). C. Gráficos tSNE que representan poblaciones celulares identificadas por Phenograph de la muestra secuenciada en un. Dr. Los gráficos circulares muestran la fracción de células dentro de cada grupo de células específico en cada grupo de pacientes. mi. El gráfico de barras muestra la distribución (porcentaje) de células de cada grupo de pacientes en cada grupo de células seleccionado. F. Se muestra un mapa de calor de la expresión del receptor para cada población de células como puntuaciones z ajustadas utilizando los valores promediados. gramo Y el h. Resumen de expresión de activación (gramo) y opresivo (h) Marcadores de histonas en monocitos de individuos sanos (n = 20), pacientes con COVID-19 leve (n = 15) y moderado (n = 11). ANOVA unidireccional con corrección de Tukey para comparaciones múltiples de b, g, h. *p<0,05, **p<0,005, ***p<0,001, ****p<0,0001.
Los monocitos leves de COVID-19 mostraron niveles elevados de H3K27Ac y H3K4Me3 en comparación con los controles, pero los monocitos moderados mostraron una expresión similar a los controles sanos. No hubo diferencia en la expresión de H3K9Me2, un marcador represivo; La expresión de H3K27Me3 aumentó en los monocitos leves en comparación con los controles y no se observó en los monocitos leves de COVID-19. Estos sugirieron una remodelación epigenética defectuosa y la posterior activación de las funciones inmunitarias innatas en pacientes con COVID-19 leve.
El perfil de expresión génica de los monocitos clásicos de pacientes leves con COVID-19 se evaluó en profundidad en relación con controles sanos. El análisis de expresión diferencial de genes reveló una regulación positiva de 422 genes y una regulación negativa de 187 genes en monocitos de COVID-19 en relación con sujetos sanos. El análisis de enriquecimiento de vías (PEA) de genes regulados al alza (en monocitos de COVID-19) mostró un aumento significativo en el metabolismo de los lípidos, el interferón (IFN) y la señalización de citoquinas.
El aumento de las firmas del gen IFN tipo I en los monocitos de COVID-19 ex vivo se confirmó mediante el aumento de la expresión del factor regulador 3 (IRF3), así como de la proteína transmembrana estimulada por IFN-γ (IFITM2), un gen estimulador de IFN (ISG). ). La PEA realizada en un conjunto de genes regulados a la baja reveló que la glucólisis era la única vía regulada significativamente en los monocitos de COVID-19. Los autores observaron anomalías en los perfiles metabólicos junto con una disminución de la activación del factor nuclear kappa-B (NFκB) y respuestas intactas de IFN tipo I en monocitos de pacientes leves con COVID-19.
A continuación, se probó la función de respuesta sensorial de los monocitos del SARS-CoV-2 ex vivo. Los monocitos de controles sanos mostraron un aumento significativo en la producción de factor de necrosis tumoral (TNF) e interleucina (IL)-10 tras la estimulación con SARS-CoV-2. Por el contrario, los monocitos de COVID-19 expresaron menos TNF que los monocitos de control, mientras que no se observaron diferencias en los niveles de IL-10. Esta expresión reducida de TNF no era específica del SARS-CoV-2, ya que la estimulación con lipopolisacárido bacteriano (LPS) o coronavirus del resfriado común (CoV) también tenía una expresión más baja de TNF.
Además, se realizó RNA-seq en monocitos activados por SARS-CoV-2 de pacientes leves con COVID-19 y controles sanos. Alrededor de 1437 y 2073 genes, respectivamente, se regularon al alza en los monocitos de COVID-19 en relación con los monocitos de control. La PEA de genes regulados al alza reveló un enriquecimiento significativo de las vías implicadas en la hemostasia y la coagulación. Las vías reguladas a la baja en los monocitos de COVID-19 fueron predominantemente funciones inmunitarias esenciales, como la señalización de IFN, la activación de la señalización del receptor de células T en las células T y las funciones inmunitarias innatas con no linfocitos.
Conclusiones
El estudio actual evaluó las características metabólicas, transcripcionales y funcionales de los monocitos e identificó varios cambios fenotípicos y funcionales en los monocitos de pacientes con COVID-19. Ex vivo, los monocitos de COVID-19 cambiaron relativamente a un fenotipo protrombótico de función inmune innata primaria al detectar el patógeno.
La base subyacente del fenotipo disfuncional (en monocitos de COVID-19) podría ser defectos epigenéticos y metabólicos. Por ejemplo, los defectos en la acetilación de histonas pueden deberse a la falta de grupos acetilo proporcionados por el producto glucolítico, acetil-coenzima A, mientras que la glucólisis se reduce significativamente en los monómeros de COVID-19. Además, se deben investigar los factores que conducen a la disfunción de los monocitos. En general, los resultados del estudio proporcionaron un mecanismo por el cual la disfunción en la inmunidad innata puede contribuir a las enfermedades por COVID-19.
*Nota IMPORTANTE
bioRxiv Publica informes científicos preliminares que no han sido revisados por pares y, por lo tanto, no deben considerarse concluyentes, orientar la práctica clínica o el comportamiento relacionado con la salud ni tratarse como información establecida.
Referencia de la revista:
- Reprogramación bíblica de la función inmune innata a la firma protrombótica en la detección de SARS-CoV-2 por monocitos en COVID-19 Alison K Maher, Katie L Burnham, Emma Jones, Laurie Bailon, Claudia Silk, Nicholas Jiang, Rafael Arguelo, Charlotte Yves Short, Rachel Quinlan, Wendy S. Barkley, Nicola Cooper, Graham B. Taylor, Emma E. Davenport, Margarita Domínguez Villar, Purxive; DOI: https://doi.org/10.1101/2022.04.03.486830Y el https://www.biorxiv.org/content/10.1101/2022.04.03.486830v1

“Defensor de la Web. Geek de la comida galardonado. Incapaz de escribir con guantes de boxeo puestos. Apasionado jugador”.